Mỗi loai tế bào lại có cấu trúc và chức năng riêng biệt để tạo nên mô, cơ quan cuối cùng là tạo một cơ thể hoàn chỉnh. Vì thế, ngoài việc nghiên cứu chức năng của một cơ quan hay mô cụ thể người ta còn nghĩ đến nghiên cứu chức năng riêng lẻ của từng tế bào để có cái nhìn chi tiết hơn về cơ thể con người.
Những bước tiến trong kĩ thuật gần đây cho phép chúng ta giải trình tự mRNA của từng tế bào (single-cell mRNA-sequencing hay scRNAseq) với số lượng lớn, độ chính xác đến từng tế bào một [5]. Kỹ thuật scRNAseq thường được sử dụng để nghiên cứu về mức độ biểu hiện gen thông qua mRNA trong nghiên cứu mô và cơ quan. Mỗi tế bào trong cơ thể tuy có thể giống nhau về kiểu gen nhưng lại biểu hiện khác nhau tạo nên nhiều loại tế bào. Các nghiên cứu sử dụng scRNAseq gần đây thường tiến hành trên não, võng mạc, tuyến tụy, tế bào miễn dịch , phôi và tế bào tạo máu [4]. Như trong nghiên cứu của Campbell và cộng sự, họ sử dụng kĩ thuật này để phân tích 20 921 tế bào từ vùng dưới đồi não chuột và đã xác định được một nhóm mới có khoảng 50 tế bào (<0,2%) [5].
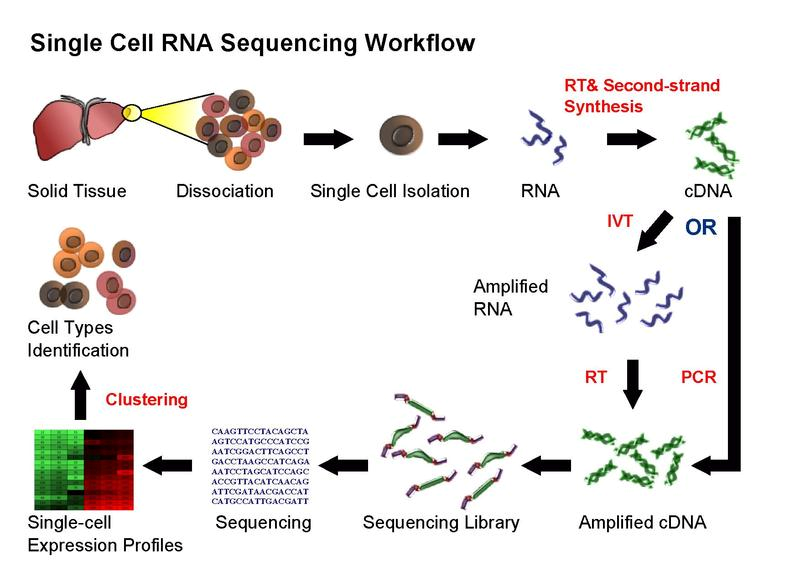
Hình 1: Quy trình của kỹ thuật giải trình tự RNA từng tế bào.
Thông thường, kĩ thuật scRNAseq theo một chiến lược khá giống nhau. Đầu tiên, một tế bào được cố định và ly giải, sau đó tiến hành phiên mã ngược mRNA để thu cDNA. Sau đó, cDNA được nhân bản qua (1) PCR hoặc (2) phiên mã in vitro rồi phiên mã ngược trở lại cDNA. Cuối cùng, lượng cDNA thu được sẽ dùng cho giải trình tự. Khác với việc giải trình tự bình thường, scRNAseq phải giải quyết hai vấn đề: cố định từng tế bào riêng lẻ và nhân bản số lượng mRNA từ một tế bào.
Làm thế nào để nuôi cấy từng tế bào riêng lẻ? Hiện nay có khá nhiều kĩ thuật có thể giải quyết vấn đề này như nuôi cấy trên giếng, trong vi môi trường lỏng và nuôi trong giọt. Các kĩ thuật này dựa trên nguyên tắc như sử dụng kháng thể (FACS), pha loãng nhiều bước, kênh dẫn vi lưu (microfluidic) hay sử dụng màng lọc (nanofilters) để có thể thu nhận từng tế bào. Một trong những kĩ thuật được sử dụng phổ biến hiện nay là microfluidic platform, kĩ thuật này sử dụng thiết kế các ô có kích thước rất nhỏ, mỗi ô chỉ chứa được một tế bào, dòng tế bào sẽ di chuyển theo một chiều nên các tế bào sẽ lấp đầy các ô.
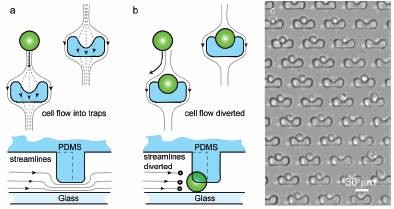
Hình 2: Kỹ thuật kênh dẫn vi lưu (microfluidic flow)
Nhằm nhân lượng lượng mRNA, có thể thiết kế nhiều loại mồi khác nhau tùy vào mục đích nghiên cứu. Mồi gồm 7 nucleotide (heptamer) thường được dùng cho khuếch đại trình tự RNA đã biết trước, sử dụng mồi ngẫu nhiên và mồi polyT để nhân bản lượng mRN trưởng thành có trong mẫu. Sau khi khuếch đại, cDNA sẽ được giải trình tự và phân tích để xem đó là gen nào và có đột biến nào xảy ra hay không?
Giải trình tự scRNAseq thường chứa các số liệu gây nhiễu, thay đổi trong các quy trình nhân bản số lượng gen hay cố định tế bào có thể có giá trị khác biệt rất lớn. Bên cạnh đó, thí nghiệm giải trình tự nhiều tế bào cùng lúc có thể thực hiện chỉ trên một thí nghiệm nhưng giải trình tự của từng tế bào thì phải cần đến hàng ngàn thí nghiệm. Lượng dữ liệu thu được vì thế cũng tăng gấp 10 đến 100 lần. Việc xử lý dữ liệu cũng gặp nhiều khó khăn do lượng số liệu lớn và thông tin gây nhiễu [8].
Vì kĩ thuật này còn tương đối mới nên đang được tiếp tục nghiên cứu, thử nghiệm và dần hoàn thiện để có độ tin cậy, sự chính xác cũng như dễ thực hiện hơn. Mặc dù còn nhiều thách thức nhưng đây sẽ là công cụ quan trọng khi nghiên cứu sâu về chức năng từng tế bào một trong cơ thể cũng như những liệu pháp điều trị trên phương diện tế bào trong tương lai.
Tác giả: Võ Thị Hạnh Đan (Đại học Khoa học Tự nhiên TP. HCM)
Tài liệu tham khảo:
4. Andrews, T.S. and M. Hemberg, Identifying cell populations with scRNASeq. Mol Aspects Med, 2017.
8. Perkel, J.M., Single-cell sequencing made simple. Nature, 2017. 547: p. 2.


